Os pesquisadores da UC San Diego introduziram o Multi-Scale Integrated Cell (MuSIC), uma tecnologia que combina microscopia, bioquímica e inteligência artificial, e revela componentes celulares até então desconhecidos que podem fornecer novas pistas para o desenvolvimento humano e doenças. (Apresentação conceitual do artista.) Crédito: UC San Diego Health Sciences
A tecnologia baseada em IA está revelando componentes celulares até então desconhecidos que podem fornecer novas pistas para a evolução e doenças humanas.
A maioria das doenças humanas pode ser rastreada até partes defeituosas da célula – um tumor é capaz de crescer porque um gene não foi traduzido com precisão em uma proteína específica ou uma doença metabólica surge porque as mitocôndrias não se ativam adequadamente, por exemplo. Mas para entender quais partes de uma célula podem dar errado em doenças, os cientistas precisam primeiro de uma lista completa das partes.
Ao combinar técnicas de microscopia, bioquímica e inteligência artificial, pesquisadores da Escola de Medicina da Universidade da Califórnia em San Diego e colaboradores deram o que acreditam ser um grande salto no entendimento das células humanas.
A tecnologia, conhecida como Multi-Scale Integrated Cell (MuSIC), foi descrita em 24 de novembro de 2021, em temperar natureza.
“Se você imaginasse uma célula, provavelmente imaginaria o diagrama de cores em um livro de biologia celular, com mitocôndrias, retículo endoplasmático e núcleo. Mas é essa a história toda? Trey Edecker, PhD, professor da Universidade da Califórnia, San Diego School of Medicine e Morris Cancer Center, disse. ”Certamente não.” “Os cientistas reconheceram há muito tempo que há mais que não sabemos do que sabemos, mas agora temos uma maneira de olhar mais profundamente.”
Ideker conduziu o estudo com Emma Lundberg, Ph.D., do KTH Royal Institute of Technology em Estocolmo, Suécia e da Universidade de Stanford.
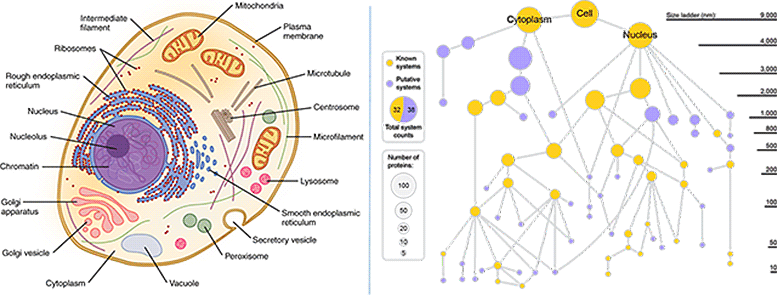
Esquerda: Diagramas de células de livros didáticos convencionais indicam que todas as partes são visíveis e claramente marcadas. (Crédito: OpenStax / Wikimedia). À direita: um novo mapa celular gerado pela tecnologia MuSIC revela vários novos componentes. Os nós dourados representam os componentes celulares conhecidos, enquanto os nós roxos representam os novos. O tamanho do nódulo reflete o número de proteínas distintas neste componente. Crédito: Universidade da Califórnia, San Diego Health Sciences
No estudo piloto, o MuSIC revelou cerca de 70 componentes encontrados na linha celular do rim humano, metade dos quais nunca haviam sido vistos antes. Em um exemplo, os pesquisadores descobriram um grupo de proteínas que formam uma estrutura desconhecida. Trabalhando com Jin Yu, Ph.D., colega da UCSD, eles finalmente determinaram que a estrutura é um novo complexo de proteínas que se ligam umas às outras. RNA. É provável que o complexo esteja envolvido no splicing, um importante evento celular que permite a tradução de genes em proteínas e ajuda a determinar quais genes são ativados e em que momento.
O interior das células – e as muitas proteínas que existem – geralmente são estudadas usando um de dois métodos: microscopia ou correlação biofísica. Por imagem, os pesquisadores adicionam marcadores fluorescentes de cores diferentes às proteínas de interesse e rastreiam seus movimentos e associações através do campo de visão do microscópio. Para analisar as conexões biofísicas, os pesquisadores podem usar um anticorpo específico para uma proteína para puxá-la para fora da célula e ver a que mais se liga.
A equipe está interessada em mapear o funcionamento interno das células há muitos anos. O que difere do MuSIC é o uso de aprendizado profundo para mapear uma célula diretamente a partir de imagens de microscopia celular.
“A combinação dessas técnicas é única e poderosa porque é a primeira vez que medições em escalas completamente diferentes foram combinadas”, disse o primeiro autor do estudo, Yue Qin, um estudante graduado em bioinformática e biologia de sistemas no laboratório de Ideker.
Os microscópios permitem que os cientistas vejam, ao nível de um mícron, o tamanho de algumas organelas, como as mitocôndrias. Elementos menores, como proteínas individuais e complexos de proteínas, não podem ser vistos através de um microscópio. Técnicas bioquímicas, que começam com uma única proteína, permitem aos cientistas chegar à escala nanométrica. (Um nanômetro é um bilionésimo de um metro, ou 1.000 mícrons.)
Mas como você preenche essa lacuna da escala do nanômetro para a escala do mícron? “Sempre foi um grande obstáculo nas ciências biológicas”, disse Edeker, que também é o fundador da Iniciativa do Mapa de Células do Câncer da Universidade da Califórnia e do Centro de Biologia Computacional e Bioinformática de San Diego. “Acontece que você pode fazer isso com IA – ver dados de várias fontes e pedir ao sistema para agregá-los em um modelo de célula.”
A equipe treinou a plataforma MuSIC AI para examinar todos os dados e criar um modelo de célula. O sistema ainda não mapeou o conteúdo da célula para locais específicos, como um esboço de livro didático, em parte porque seus locais não são necessariamente fixos. Em vez disso, as localizações dos componentes são fluidas e mudam de acordo com o tipo de célula e a situação.
Edecker observou que este foi um estudo piloto do teste MuSIC. Eles analisaram apenas 661 proteínas e um tipo de célula.
“O próximo passo óbvio é infundir toda a célula humana e, em seguida, passar para diferentes tipos de células, pessoas e espécies”, disse Edecker. “Em última análise, podemos ser capazes de compreender melhor a base molecular de muitas doenças, comparando o que é diferente entre células saudáveis e doentes. “
Referência: “Mapa multiescala da estrutura celular incorporando imagens de proteínas e interações” por Yu Chen, Edward L. Hotlin, Casper F. Winsens, Maya L. Guztaila, Ludivine Wachul, Marcus R. Kelly, Stephen M. Blue, Van Zing, Michael Chen, Leah F. Shaffer, Catherine Lecon, Anna Backstrom, Laura Pontano Weitz, John J. Lee, Wei Ouyang, Sophie N. Liu, Tian Zhang, Erica Silva, Jisoo Park, Adriana Petty, Jason F. Kreisberg, Stephen B . Gigi, Jianzo Ma, J. Wade Harper, Jane W.U., Dennis LJ Lafontaine, Emma Lundberg e Trey Edecker, 24 de novembro de 2021, temperar natureza.
DOI: 10.1038 / s41586-021-04115-9
Os co-autores são: Maya L. Guztaila, Marcus R. Kelly, Stephen M. Adriana Pitea, Jason F. Kreisberg, UC San Diego; Edward L. Hotlin, Laura Pontano Weitz, Tian Zhang, Stephen B. Gigi, J .; Wade Harper, Harvard Medical School; Casper F. Winsnes, Anna Bäckström, Wei Ouyang, KTH Royal Institute of Technology; Ludivine Wacheul, Denis LJ Lafontaine, Université Libre de Bruxelles; e Jianzhou Ma, da Universidade de Pequim.
O financiamento para esta pesquisa veio, em parte, dos National Institutes of Health (Grants U54CA209891, U01MH115747, F99CA264422, P41GM103504, R01HG009979, U24HG006673, U41HG009889, R01HL137223, R01HG004654 Research420171HGM103504, R01HG009979, U24HG006673, U41HG009889, R01HL137223, Fundação R01HG0046542017 Sueco, Grant242027201717 Sueco). , a Universidade Livre da Bélgica em Bruxelas, o Programa Europeu Conjunto para Doenças Raras, a Région Wallonne, a Internationale Brachet Stiftung e o Procedimento COST Epitran (bolsa CA16120).
Divulgações: Trey Ideker é cofundador do Conselho Consultivo Científico e tem uma participação na Data4Cure, Inc. Jin Yu é cofundador e membro do conselho do Conselho Consultivo Científico, acionista e consultor pago da Locanabio e Eclipse BioInnovations. Yu também é professor visitante na Universidade Nacional de Cingapura. Os termos desses acordos foram analisados e aprovados pela University of California San Diego de acordo com suas políticas de conflito de interesses. Emma Lundberg é membro dos Conselhos Consultivos Científicos em Mapping Biology, Nautilus Biotechnology e Interline Therapeutics, e tem interesses iguais em Mapping Biology. J. Wade Harper é cofundador do Scientific Advisory Board e tem participação na Caraway Therapeutics. Harper também é o conselheiro científico fundador de Terapias Interdisciplinares.